番茄中广泛的结构变异对基因表达和性状改良中的作用
本文通讯作者来自冷泉巷实验室

https://www.cshl.edu/research/faculty-staff/zachary-lippman/
研究植物开花和花的发育过程
利用遗传学、基因组学和基因编辑技术研究植物分生细胞发育成花中的分子机制
约翰霍普金斯大学 Michael Schatz
主要对基因组结构和功能进行研究,针对大规模的测序数据开发新的算法
Abstract
结构变异是一种基因组间的序列差别,构成了作物改良和驯化的基础。由于短序列测序技术的限制,分析SVs差异的程度以及量化对性状的影响程度,一直非常困难。通过对100个番茄野生株系使用nannpore测序,与标准基因组比较后鉴定到238490个SVs。结合这些panSv数据以及组织了14份新的参考基因组,表明不同基因型材料间存在大规模的混合,大量的SVs与基因区、cir-regulation区存在交集。与此同时数百个SV-gene表现出基因表达水平的改变,这可能影响数量性状位点。通过结合数量性状和基因组编辑技术,作者发现,多个SVs能够改变基因的剂量和表达水平,从而导致口感、大小、以及产量性状的改变。四个SVs影响3个相关的转录因子,使得一个产量性状的改变。该研究突出了SVs在基因型到表型关系中的作用,强调了在作物改良中的重要性和实用性。
背景
作物的表型变异是受到来自野生祖先种种遗传变异的影响,同时人类的选择和保持一些突变集合同样会影响作物的表型。主要的变异是数量遗传变异,受到多个微效基因的控制,因此现在遗传学的目标主要是解析特定基因和变异对数量性状的影响。人们通过高通量的短读序列测序,在大多数农作物中鉴定了自然遗传变异,但是这种方法只能够使用SNP、或者小片段indel来描述遗传变异,往往忽略了其他因素,因此会带来一定的偏差。最近10几年来的研究,人们发现SVs(大片的的插入、缺失、重复、染色质重排),对植物的进化以及一些重要的农艺性状产生巨大的影响,例如根尖结构、开花时间、果实大小、抗逆性。与SNP相比,SVs往往会造成cis调控区域产生大规模的扰动,进而有可能导致基因表达发生改变,表型发生变化。SVs同样可以通过直接改变基因的拷贝数,来改变基因的表达水平,尽管SVs如此重要,但是通过短读测序鉴定SVs非常困难,使得大多数SVs没有被鉴定出来,并且它们的分子机制和对表型的影响被隐藏起来。
全长Nanopore 测序,使得我们能够广泛的研究,群体规模的SV图谱,鉴定出SVs之间的差异,结合表达量的数据和基因组编辑技术,使得我们能够快速而直接的推断出SVs的分子机制和对表型的影响。在此,我们提供了番茄中最全面的 panSV图谱,并且研究它在进化、循环、数量遗传、育种中的作用。
作者使用nanopore测序了包含野生种和栽培种的100份材料,并且鉴定了SVs。对超过200000个SVs序列进行分析发现,主要是转座子和重复元件。
对SVs的分布进行分析,发现群体水平内存在广泛的基因渗入
RNA测序分析表明,SVs通过影响cis区域、编码区域来影响基因的表达
重头组装的14份材料的基因组,使得我们能够更好的解析基因组的复杂性。
结合CRISPR-Cas9技术,表明了基因的剂量效应和表型之间的关系。
结果
1.结合long-read测序,构建PanSV genome
收集了具有代表性的800份番茄材料,使用二代数据鉴定了SVs,选择了其中51份来自早期和现代栽培品种,并且包含了最多的SV信息;同时选择了49份野生种。这些品种主要分为:
SP CHE GAL
SLC
SLL
对100份材料进行40X的long read测序,将序列比对到标准参考基因组上鉴定SVs,筛选>30bp的SVs并且合并和作为panSV genome,经过确认发现大多数SVs在短读测序中没有鉴定到。

B 图 单个材料鉴定的SVs范围在1928~45840之间,其中在野生种材料中包含有最多的SVs,在SVs的种类里出现最多的是插入与缺失,也有少量的易位于重复。
C图 基于SVs的有无,反应这些材料间的进化关系,可以发现在栽培种和半驯化品种中有一些栽培种与半驯化品种聚类在一起,说明他们之间有渗透。

D图 比较每个材料中SVs的数目,发现在野生种中SVs数量最多,同时栽培种中SLL最少,这样驯化过程中遗传资源的丢失是吻合的
E图 表明这100份材料的SVs仍旧有许多的SVs没有被发现
F图中可以发现很多SVs只在少数几个材料中被鉴定到。
G图 评估SVs的长度分布,发现大多数的SVs是比较小的,只在30~50bp

2.组装14份高质量的番茄参考基因组
结合panSV genome和其他的genome信息,作者选择了14个差异比较大的材料,进行了基因组的组装和注释。组装的新的基因组叫MAS2.0,与最近发布的ITAG进行比较,发现了一些新的“pan-genome“ gene。
3.SV在材料间的分布,表明不同材料间发生看遗传渗透
A图 中在栽培种SLL中SVs热点区域,同时由于野生材料SP具有更多的SVs,SVs热点区域可能反应的是品种驯化过程中,遗传资源从野生种中渗透到栽培种中的。在番茄的育种中,通过将一些抗病、理想性状从野生供体中引入到SLL中。
从B中可以 ,看到有5个SLL材料中共有的渗入区间,其中两个是M82 and EA02054 ,这两个材料在5号染色体与11号染色体还有大的渗入区间,这也解释了在构建的系统发生树中,这两个材料与栽培材料隔得远

D图 对11份SLLs品种的育种历史进行考察,真菌性镰刀菌病,在1930年首次出现,在1930~1980年,从SP材料中导入了I I2 I3个抗性基因。对这些基因进行分子克隆,发现它们主要位于11号染色体两端的臂,与UFL 的渗入区间有交集,这个区间内部还有一些其他的抗性基因。Fla.8111B 材料同样包括了这些抗病基因,不过没有对应的渗透区间,说明这些材料经过了纯化选择,将这些区间给过滤了,只保留下了抗性基因。

与基因有关的SVs,对表达有影响
SVs通过改变基因序列、基因拷贝数、cis调控序列的位置或者组成,来改变富集基因的表达。
有50%的SVs与gene或者两侧的调控区存在交集,在34075个注释的基因中,有95%的基因附近存在至少一个SVs,并且主要是在cis调控区域。

为了探讨SVs对gene表达的影响,选择23份材料进行RNA测序,共得到21,156 份gene-SV对,发现很多gene存在显著的改变。其中将近一半的SVs影响编码区的,表达发生显著的改变。
cis-eQTL能够解释了群体中造成表达差异变异的很大部分,作者使用分类模型通过基因表达水平的变化,预测附近SVs的有无,量化基因表达水平的变化与SVs间的相关程度。从E图中可以通过AUROC指标,发现分类器在对编码区的SVs进行预测的效果最好,总的来说,SVs能够轻微的影响基因的表达。

新组装的基因组发现了多个挥发性的单倍型基因
通过新组装的MAS2.0、表达谱的数据,帮助我们找到一些影响数量性状的变异位点。通过GWAS,已经鉴定出的具有水果挥发性香气的QTL,但很少一部分进行了功能研究。其中一个QTL编码水杨酸甲脂,对消费者的吸引产生负面影响。
还有一个E8基因,在水果的成熟过程中,负调控乙烯的生物合成;敲除E8基因后又导致一些不好的挥发物质的积累。由于当时基因组中E8两侧存在gap,所以没有其他的变异被检测到。
A图 另外一个研究对NSGT1和NSGT2突变后,造成愈创木酚的积累,NSGT1少量表达被认为是没有功能的,在成熟过程中NSGT2的上调表达,使得愈创木酚转化成三糖苷,从而预防了愈创木酚的挥发。为了研究NSGT1与挥发性物质的关系,检查了这个基因的物理位置。B图 发现在一起的基因组中这个基因的注释不完整,而在新组织的MAS2.0中,一共发现了5种组合C图

经过分析发现这些基因E8、NSGT1、NSGT2的变异构成了这个愈创木酚的变异。使用300份表达谱数据和代谢数据,计算了每种单倍型的关联程度。其中IV和V是在早期驯化品种SLCs中出现的。
E图中有5个V型材料,NSGT2没有检测到表达
在图F中,IV和V在统计上没有差异,但相比于I II III都变现出更高含量的愈创木酸
G图使用一个新的群体包含SP、SLC进行关联分析后与F图结果类似
H图使用两个SLC构建的F2群体,表明E8和NSGT1的缺失都导致愈创木酸含量的提高
这个结果表明高质量的基因组,能够帮助我们解决一些复杂的单倍型基因,同时从QTL分析中解释基因型与表型

细胞色素P450基因与果实大小
与果实大小有关的QTL已经被鉴定出来,其中最显著的fw3.2,是位于P450 SlKLUH启动子区域的一个SNP,这个SNP被认为能够提高SIKLUH基因的表达。
通过将这个SNP位点与panSV genome比较发现这个地方有串连重复,其中有两个SIKLUH的相同拷贝,fw3.2 dup。
通过分析这个重复基因的有无对内部三个基因表达的影响发现,在存在重复基因时,表达量更高;同时为了探究到底是启动子区域SNP还是这个重复基因对果实大小造成影响,作者构建了F2群体,通过分离重复基因,其他QTL仍旧固定,D图中发现共分离。而SNP的有无对果实大小没有影响。

作者认为fw3.2通过基因拷贝数的增加,造成剂量效应,从而使得果实变大。通过CRISPR-Cas9,造成大片段和小片段的缺失,进行PCR发现这些植株表型发生明显的改变

在T0-1植株中表型没有很大变化,都存在野生型和突变的等位基因,利于我们研究剂量效应。进行杂交,其中野生种LA1589具有2个拷贝,在A组杂交中参数3个拷贝的F1,在B组中只有一个有效的变异,在图H、I中它们的花序、果实大小都有明显的差异

这个结果也表明panSV genome 结合CRISPR-Cas9编辑揭示了基因的剂量效应导致果实大小发生改变
四个SVs造成的“无果柄”育种
无果柄是番茄收获中重要的一个性状,使得果实从花卉中分离,研究表明它是由转座子的插入导致一个转录因子Js的表达下调。但是在引入无果柄后,又会导致过多的花序分支导致果实产量的下降。图A
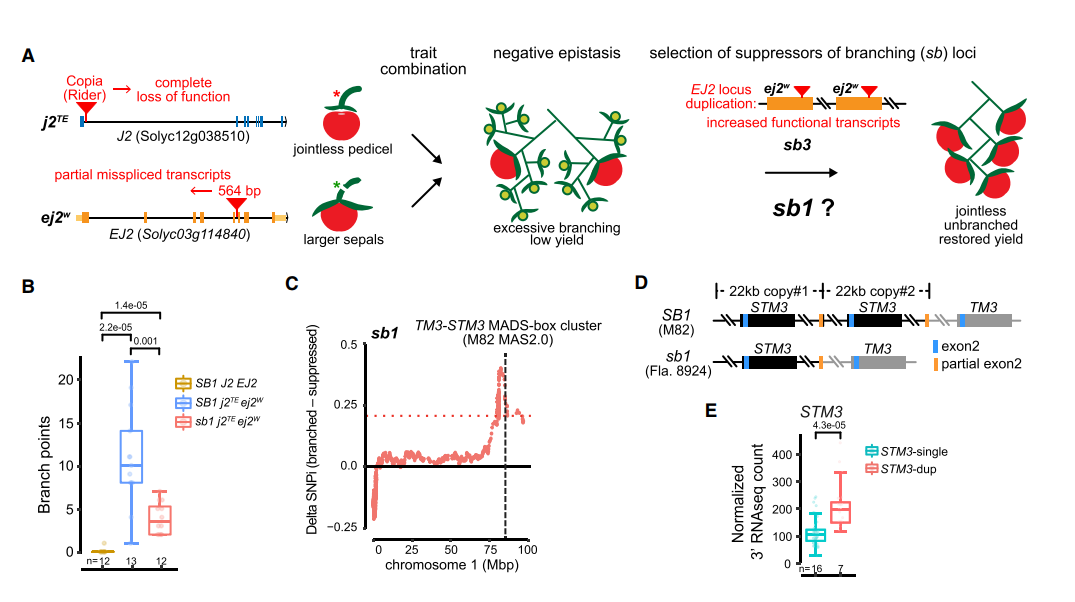
育种家们为了克服这个问题,找到了两个QTL位点 sb1和sb3,能够恢复正常花序的表型。
作者发现sb3是一个包含ej2的83kb重复,通过剂量效应弱化由于转座子插入导致的ej2表达水平的下降。
sb1则是一个抑制分支产生,定位到1号染色体6Mbp的区间;发现两个基因STM 、STM3;在分子数存在差异的两个亲本中SMT3在分支数比较多的材料里高量表达。这个基因的序列和调控区域没有明显的变化,而在原来的基因组中这里有gap,在MAS2.0中,揭示了这个地方存在22kb的串连重复拷贝,图D
为了探究这个低拷贝的SMT3基因是否就是我们要找的sb1 QTL信号,作者使用CRISPR-Cas9技术打断了STM3和TM3基因座。两种CR-1和CR-del,两个突变体在花序表型上没有很大的变化 图F;在与多花序品种杂交产生的F2群体中考察花序的数目,发现SMT3敲除后,花序分枝发生减少
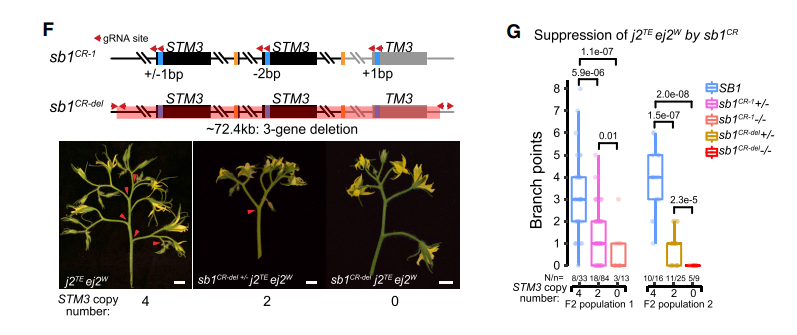
通过分析发现STM3作为新的调控花序发育的基因,对sb1的分析表明4个SVs影响了三MADS-box genes 的表达,从而影响花序的发育和果柄,进一步说明结构变异对基因功能研究的重要性
总结
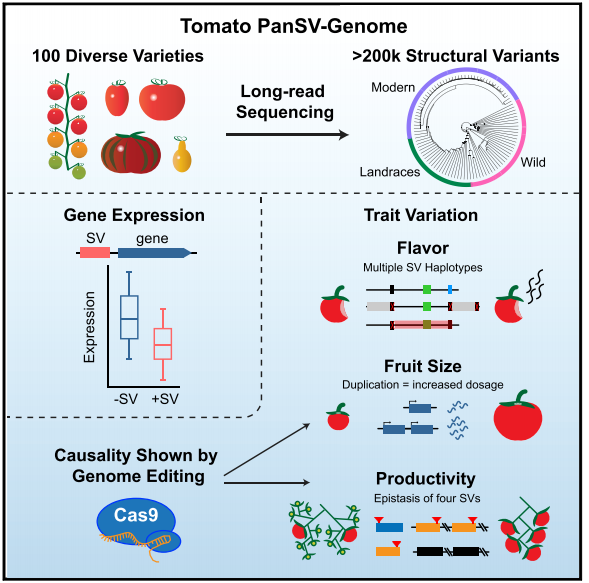
构建了植物panSV genome 数据,表明SV和基因差异表达相关
对3个具体功能的研究表明,结构变异是导致表型差异的原因
全面的植物结构变异数据,为精准的育种提供帮助
利用基因编辑技术验证SV(拷贝数变异)的功能,对其他作物的研究具有指导意义
参考
Major Impacts of Widespread Structural Variation on Gene Expression and Crop Improvement in Tomato
Last updated