可变剪接的研究进展及展望
1.背景
可变剪接的类型,和形成机制
在动植物中的研究进展
可变剪切的作用,增加蛋白组的复杂性,调控基因表达
全长转录组测序有助于鉴定AS
2.影响可变剪切的因素
基因转录过程中的动力学耦合
表观遗传(DNA甲基化、组蛋白修饰、TE)在基因转录调控中的作用
lnRNA 、miRNA
3.可变剪切的研究思路
可变剪切在基因家族和同源基因间的进化
结合sp-QTL、共表达网络,在群体水平研究可变剪接,分析组织不同发育阶段
结合基因编辑技术,对可变剪接的功能进行验证
4.在研究棉花、或者多倍体植物带来的思考
文献解读
1.背景部分
基因可变剪接的类型
Post-transcriptional Processing of mRNA in Neurons: The Vestiges of the RNA World Drive Transcriptome Diversity
蛋白质编码基因的转录受到启动子的调控,在包含启动子的转录起始区域TSS,通过招募转录因子、RNA聚合酶II;起始基因的转录。在转录过程中,包含多个外显子的pre-mRN通过剪切机制去除内含子,连接两端的外显子;而在这个过程中通过可变剪接包含或者排除特定的exon形成不同类型的转录本;在细胞核内,pre-mRNA还经历了5’加帽,3‘端多聚腺苷化过程。因此同一个基因可以通过选择不同的转录起始位点、转录终止位点、不同的剪接形式,产生多种mRNA isoform;而我们今天主要探讨的就是AS在基因转录过程形成、以及对应的调控机理。

对于一些剪切位点的选择,将可能导致IR、ES等剪切事件;使得单个基因产生多个isoforms
在基因的选择性剪接中,最主要的选择剪切模式包含以下四种:
IR 内含子保留
ES 外显子跳跃
AltA 可变的5端
AltD 可变的3端
在人类中高达92%-94%的多外显子基因经历AS事件,而植物中有21.2%~63%的基因经历AS事件。
在人类和动物中SE事件所占所有AS 事件的40%,而在植物中IR事件占据AS事件的40%;
这可能是由于动物中的大部分内含子都比植物中的内含子短,导致剪切复合体识别的差异:
内含子识别模式
外显子识别模式

Perspective on Alternative Splicing and Proteome Complexity in Plants
对于多细胞生物来说,在正确的时间、正确的位置,产生正确的蛋白质对于生物的生长发育至关重要;蛋白质的合成受到多种因素的调节,而其中基因的转录调控是其中重要的一种。
AS翻译出具有功能的蛋白,增加蛋白质的多样性,响应环境刺激
植物在胁迫条件下,基因通过可变剪切调节转录组的多样性和蛋白组的复杂性来响应外界的变化。在人类的研究中表明AS的存在显著的增加了蛋白质的多样性,但是在各个蛋白组组学的研究中很少检测到由AS isoforms编码出差异的蛋白;这个结果相互矛盾。
AS同样能够导致编码框的偏移,使得翻译出截断的蛋白质;这些蛋白质可能仍旧存在一些功能模块,但是缺失一些功能域或者蛋白质修饰位点。可能会与正常蛋白有竞争效应。
质谱仪在区分AS isofrom蛋白方面的敏感性比较差
并不是所有的AS isoform都具有生物学功能

Complexity of the Alternative Splicing Landscape in Plants
AS的另一个作用:因为AS产生的IR事件大多数是NMD的靶标,通过NMD将AS转录本降解,调节稳定转录本和un-stable转录本的数量。因此AS能够影响基因的转录丰度。
基因的转录和翻译都需要消耗巨大的能力,尽管如此植物在非胁迫条件下还是转录出大量的AS isoform。如果AS的作用是翻译出响应胁迫的蛋白质的话,植物就没有必要在胁迫降低时,仍旧把大量AS的转录本进行翻译浪费能量。
好在,细胞质中存在存在一种无意义介导的降解机制(NMD);通过识别提前终止的密码子,将新和成的mRNA降解;调节基因的转录水平、或者快速响应外界环境的刺激。

RNA sequencing: the teenage years
1.随着三代全长转录组测序技术的发展和应用,使得我们可以直接对整个转录本进行测序,避免了由于short read在转录本组装过程中不准确的问题,更加准确的鉴定基因的AS事件,甚至于可以借助Direct-seq 技术直接对RNA进行测序(不经过反转录,从而保守了mRNA上的修饰)。

借助于测序技术的发展
在一些物种中对于可变剪接的研究也变得更加的准确与方便。在高粱中首次使用PacBio全长转录组测序技术对AS、APA进行分析,与已有的注释相比Iso-seq显著的改善了基因的注释,找到了更多的剪切事件。
使用Iso-seq测序,比较了玉米和高粱在11个组织中AS的差异,表明亚基因组在isofrom 的产生上不存在差异。同时与旧的基因相比,年轻的基因拥有更少的转录本数、基因、ORF长度也更短;
在油菜中结合Iso-seq和Illumina 二代测序技术对全基因组的AS事件进行鉴定表明,31591个AS事件坐落在ORF区域,并且可能导致CDS编码框的改变,大约69%的AS事件是组织特异性的。
2.影响可变剪切的因素
剪切和RNA共转录部分

1.Splicing and transcription touch base: co-transcriptional spliceosome assembly and function
RNA的剪切主要发生在转录过`程中,通过将剪接复合体逐步的组装到新生的RNA上,从而实现催化剪接功能。
并且剪切复合体与RNA聚合酶II的物理位置,非常接近说明剪接与转录发生的时间很接近;
转录延伸和暂停会影响剪切复合体识别保守的位点,影响剪切复合体的组装;一些表观因素,DNA甲基化、组蛋白修饰等会影响转录动力学,改变转录的延伸速率。
转录后的剪切
Post-transcriptional splicing of nascent RNA contributes to widespread intron retention in plants
富集结合在染色质上的RNA,进行测序可以同时在全基因组水平检测转录本的剪切状态、RNA聚合酶II的位置、多聚腺苷酸化;分析发现大量腺苷酸化的mRNA包含有未剪切的内含子,而这些未剪切的内含子在细胞质中的mRNA是缺失的;这些转录后剪切的转录本受到剪切蛋白PRMT5 and SKIP 的调控;

核小体定位
Nucleosome positioning as a determinant of exon recognition
外显子处的核小体密度显著性的高于内含子,通过将外显子剪切水平与核小体的分布进行相关性分析,说明核小体在染色体水平上的位置定义了外显子。同时核小体与exon的在长度上接近,维持在147bp作用,并且exon受到强烈的选择压,认为exon子处富集核小体的作用是保护这段用功能的DNA序列

DNA甲基化、转座子
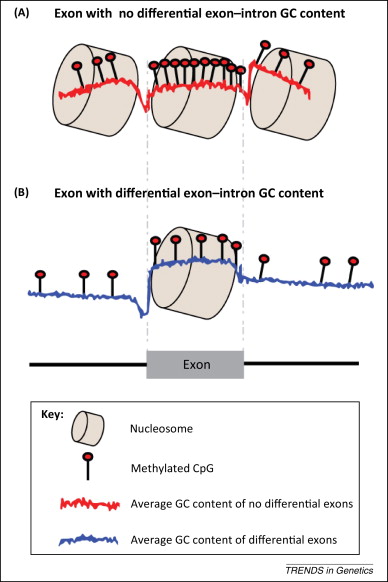
The alternative role of DNA methylation in splicing regulation
(筛选那些exon和两侧intron在GC含量上不存在差异的exon)发现外显子相比于两侧的剪切位点处有着更高水平的甲基化,
DNA甲基化调控AS的两种机制:
CTCG结合因子和CpG甲基化结合蛋白MeCP2影响RNA聚合酶II的延伸
通过HP1异染色质蛋白形成蛋白桥,招募剪切因子进行剪接
组蛋白修饰
Regulation of alternative splicing by local histone modifications: potential roles for RNA-guided mechanisms
组蛋白的甲基化和乙酰化被广泛的研究,组蛋白修饰在DNA转录过程中起着重要的作用

H3K9me3 作为异染色质结合蛋白HP1结合位点,招募剪切因子调控可变剪接,一般乙酰化的组蛋白修饰标志着更加开放的染色质区域,因此组蛋白乙酰化区域,改变局部的RNA聚合酶II的延伸速率使得第18个exon被skip,没有被剪切

总结

总的来说,转录和剪切同时进行的动力学效应,容易让人想到表观遗传通过影响染色体结构,RNA聚合酶II的延伸速度;从而参与到剪切调控。
染色体水平上的改变,例如核小体定位、组蛋白修饰、DNA甲基化,以及RNA二级结构、和反式调节因子SREs共同调控基因的剪切。因此在研究AS的时候就非常又必要整合多个组学的数据,解析植物中的剪切调控机制。
3.可变剪切的研究思路
AS在和gene duplicate
Divergence of duplicated genes by repeated partitioning of splice forms and subcellular localization
基因的duplicate 和AS都有助于蛋白组的多样化,因此植物在多倍化过程中;duplicate基因的AS在进化中是否会有差异。
例如在拟南芥等多种被子植物中分析APX(抗坏血酸过氧化物酶),重复基因中转录本的情况。

Genome-Wide Association Analyses Reveal the Importance of Alternative Splicing in Diversifying Gene Function and Regulating Phenotypic Variation in Maize
在包含368个玉米自交系群体中,进行sQTL(剪切数量性状基因座)分析,在定位到的大多数sQTL,不同基因型的主要转录本的表达ratio没有改变。同时转录本和mRNA总表达的差异是使用不同的顺式元件调节的结果。(sQTL和eQTL进行比较 figure 8A);说明仅仅从基因总的表达量来研究表型,仍旧还有很多转录本的遗传变异没有鉴定到。

构建基因共表达网络
Co-expression networks reveal the tissue-specific regulation of transcription and splicing
大多数基因共表达网络,往往是在单个组织中基于基因的总的表达量构建的genee-gene间互作的网络。在这篇文献中,基于基因的表达量和转录本的相对表达量在GTEx项目多个组织中同时用于构建基因的表达网络,进一步的加深了对于基因转录调控和基因转录后调控的理解。

Genetic Control of Expression and Splicing in Developing Human Brain Informs Disease Mechanisms
大部分基因组的研究鉴定到的一些致病位点主要位于位于非编码区、高度连锁区域。这使得对应疾病的致病机理和功能解析非常困难;由于这些非编码区的变异主要富集在一些调控区域、保守区域,它们就可能起到调节基因表达和剪切的功能。
eQTL在不同组织、不同时期间存在差异;如果在调控细胞发育的关键节点,调控区域发生变异将会导致疾病的发生。作者在原先的基础上搞了一个更大的群体,同时加上了sQL分析;对比了产前和成年人大脑的遗传调控,表明两个时期间存在大量的差异。eQTL和sQTL对于疾病的都有非常重要的贡献,并且表现出非重叠的模式。将eQTL、sQTL分别与TWAS进行整合,鉴定到一些未发现的新的机制。
sQTL、eQTL以及共表达网络在脑组织不同发育阶段的研究。
作者通过对201份脑组织样本分析
位点。通过TWAS将eQTL和sQTL与表型进行关联分析,鉴定到数十个候选的致病基因,阐述精神分裂症疾病在脑组织不同发育阶段的共有和时期特异性的调控机制。
基因共表达网络分析,表明精神分裂和自闭症影响不同发育基因的共表达module
研究表明在大脑的不同发育阶段,调控基因表达和剪接的变异存在很大的不同;并且剪切变异所带来的疾病风险比表达变异大得多的多。
1.数据来源
对233个产前大脑组织进行RNA-seq和genotyping,进过质量过滤和标准化最终得到201份大脑组织的RNA-seq和genotyping样。
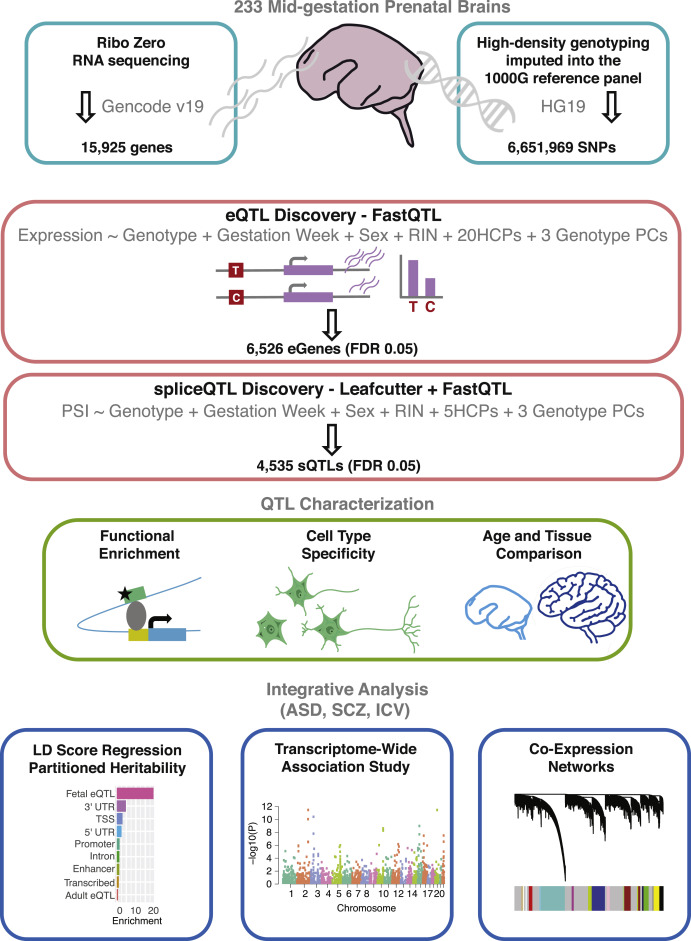
2.eQTL的鉴定
鉴定到7,962个eQTL位点其中20%的eQTL在转录起始位点的10KB范围内,表明启动子区域的变异会对应基因的表达有着巨大的影响,并且eQTL位点与其他调控区域例如ATAC、Hi-C存在显著性的重叠,表明eSNP既存在近端也存在远端区域调控靶基因的表达。对eQTL对应的eSNP进行注释,表明这些eSNP主要位于TSSs,启动子、增强子等调控区域。
探讨eQTL位点附近是否会有TF或者DNA binding蛋白的结合位点,显著的富集一些与大脑发育相关的转录因子和DP。
挑选了其中的一个转录因子CHD8,和CHD8的结合的eSNP,发现这个结合位点eSNP不同的基因型,会导致靶基因SRR表达水平的改变,同时敲除CHD8转录因子得到了同样的结论,说明CHD8转录因子可能通过结合到eSNP附近,调节基因的表达

3.sQTLs的鉴定 RNA剪切的异常同样会导致疾病的发生,同时在人类脑组织中还没有系统性的分析基因的剪切调控,因此进行了sQTL的分析。在鉴定到的4,635个sQTL其中64%的sQTL位于基因body区域,与eQTL相比sQTL在启动子和转录区域更加富集。
为了评估鉴定到的sQTL的真实性:
对RNA结合蛋白(剪切调节因子)在sSNP出的富集进行分析,发现36种实验证明的RBP显著性的富集在sSNP区域。对TRMT1这个基因进行验证,发现SRRM4(RNA结合蛋白在超表达会改变内含子的剪切比率PSI),同时不同基因型间这个内含子的剪切比例存在差异。
比较eQTL与sQTL靶基因的重叠情况:
有67%的sGene会影响基因的表达,而仅仅只有20%的eGene会影响基因的剪切,同时同一个基因的eSNP和sSNP在存在一定间隔,说明基因的表达调控和剪接调控受到独立的监管。

4.eQTL效应与组织特异性
不同组织中表达会发生变化,因此在进行eQTL分析时也会鉴定到不同的eSNP。作者分析了不同组织间eQTL效应的大小发现,在多个组织鉴定到的eQTL对基因表达量的效应显著性的低于组织特异性的eQTL效应;这说明组织特异性的调控模式的改变相比于蛋白质编码区域的改变,对生物的破坏性更小。
之前对于染色质开放性的研究,表明调控SCZ(精神分裂症)的遗传变异主要富集在大脑发育早期的调控区域,比较了不同发育阶段中的QTL对SCZ的遗传力,比较发现不同时期的QTL对于SCZ都有特定的解释力,同时又相互补充。
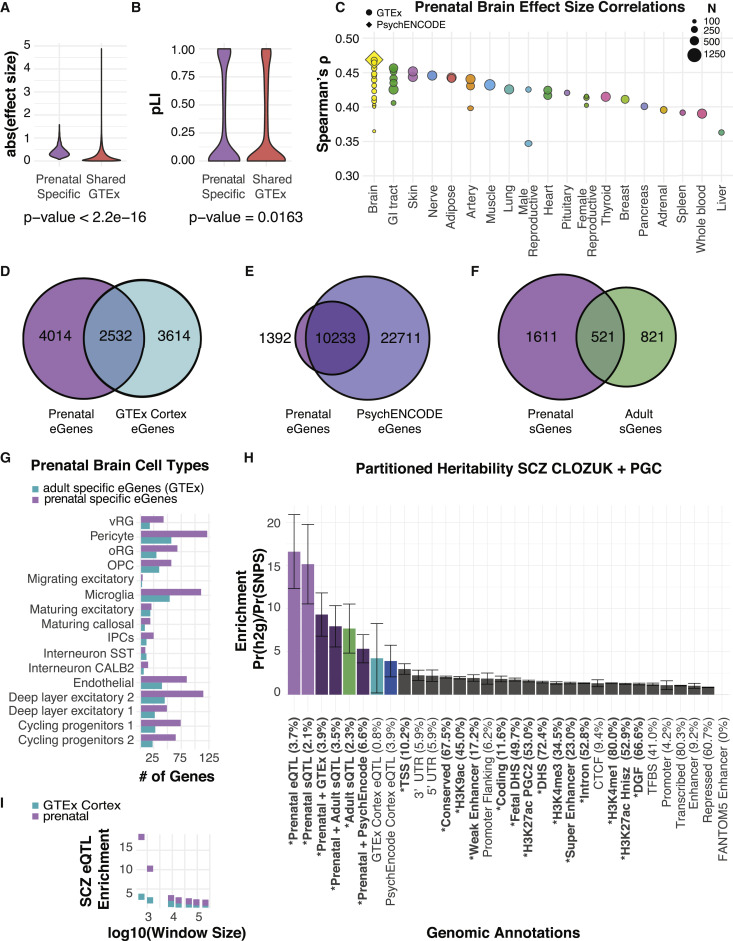
5.比较不同发育阶段的eQTL
在大脑发育中期和GTEx项目成年人的大脑中,鉴定到2532个重叠的eGene,与另外一个更大的项目,发现仍旧有1393个在大脑发育早期的eGene;同时对sGene进行比较,表明许多剪切事件是时期特异性的,可能对组织更大。
6.eQTL与表达调控网络
使用基因的表达量,构建加权共表达网络,鉴定到了19个基因module,其中的6个module基因显著的富集在一些与大脑发育相关的GO通路当中。将共表达中每个module中的eQTL与GWAS进行QQ-plot,发现一些显著性的module。当与共表达模块整合在一起时,可以发现eQTL可能能够通过调节一些具有相似功能的基因来改变基因的表型。

7.整合cis-QTL与GWAS鉴定新的候选基因
为了鉴定与疾病相关的候选基因,作者整合cis-eQTL、cis-sQTL和GWAS的数据进行TWAS分析,鉴定其表达量、剪切与表型相关的基因。一共鉴定到62个基因和91个introns与精神分裂症存在显著关联。为了减少由于连锁或者基因多效性带来的假阳性,作者使用不同方法进一步对候选基因和intron进行筛选,最终鉴定到8个潜在的致病基因和6个intron.
在不同时期鉴定到的候选基因、intron进行比较和功能验证。

结合基因编辑技术对可变剪切进行验证
CRISPR-Mediated Base Editing Enables Efficient Disruption of Eukaryotic Genes through Induction of STOP Codons
传统的CRISPR通过打断DNA双链,实现对基因的敲除;单碱基编辑通过识别四种密码子(CAA、CAG、CGA、TGG)实现碱基替换引入终止密码子,同时可以使用RFLP在群体和细胞中快速检测iSTOP的编辑效果。在8种真核生物中97~99%的基因都能找到iSTOP的靶标
CRISPR-SKIP: programmable gene splicing with single base editors (改变内含子和外显子边界保守序列,影响剪切复合体的组装实现对可变剪切的控制与调节)


4.在棉花或多倍体物种中的应用
在进化和多倍化的角度,分析基因的AS与基因加倍
AS在亚基因组间的差异,同时与染色质、表观遗传等多组学数据结合
AS在在纤维不同发育阶段的动态变化
结合eQTL、sQTL在群体水平,分析基因的转录调控和剪接调控对于表型的作用
AS的进化
Last updated