On the existence and functionality of topologically associating domains
Hi-c背景
染色体是有DNA和组蛋白共同组成的,从染色体的一级结构(序列水平上),到四级结构(超螺旋);细胞的DNA最终被压缩在微小的细胞核之中。随着测序技术的发展,生物基因组中的序列和调控元件的注释信息相继发布,人们发现这些调控元件和基因在基因组中的分布是相对离散的;而这种离散的分布不能够很好的解释生物中基因的复杂调控机制,这也就推动了对于染色体空间结构的研究。

测定染色体三维空间结构的技术的发展
3C
用于测定染色体中特定点到特定点的交互
using spatially constrained ligation followed by locus-specific polymerase chain reaction (PCR)
4C
测定一个点到多个点之间的交互
Adaptations of 3C have extended the process with the use of inverse PCR (4C)
5C
测定多个点到多个点之间的交互
multiplexed ligation-mediated amplification
Hi-c
全基因组范围内捕获染色质的交互
Hi-c测序原理
第一篇science
Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome
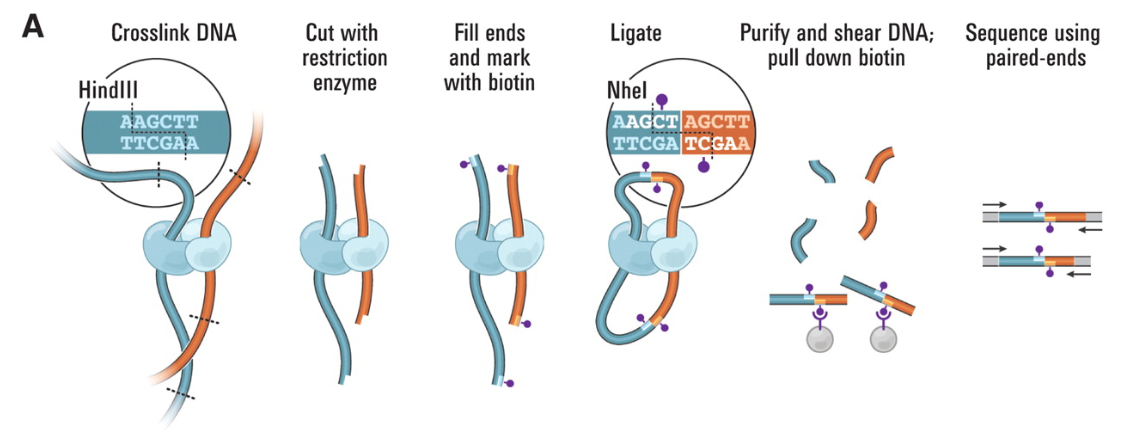
1.使用甲醛将染色体之间交联的状态进行固定
2.用限制性酶切DNA片段,留下5'粘性末端
3.使用带生物素残基的碱基对5'粘性末端进行填充;形成平末端有利于交联DNA片段之间的连接
4.发生互作的DNA片段在连接处有生物素标记,通过使用链霉素和磁珠对这些交联片段进行富集
5.建好的文库上机测序
染色质交互区域 contact domain
在Hi-c的研究中将染色质的交互分为了以下三种
1.TADs
nature中的TAD介绍
Topological Domains in Mammalian Genomes Identified by Analysis of Chromatin Interactions
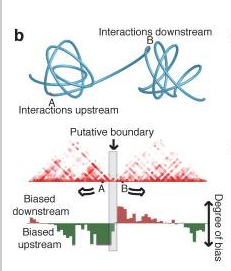
通过定义100Kb的bin去扫描时整个染色体时,出现一些非常高的交互区域,被称为TAD,紧接着一段比较短的片段,这个短片段内的交互戛然而止,因此作者猜想这种交互作用的突然变化,可能是TAD的边界区域,将基因组内的TAD分隔开来。
boundaries区域富集了绝缘子蛋白CTCF,管家基因、tRNA、SINE反转座子等
2. subTADs
随着测序精度的提高,人们发现在TADs区域还存在着更小的亚区,subTAD之间也同样被boundaries分隔,不过隔离的强度弱一些,并且在不同细胞类型中表现出cell-type-dynamic;说明subTADs与TAD相比可能存在一些其他的结构或者功能

3. compartment domains
染色体域的形成假说 Loop extrusion
Corner-dot
某一个点有着显著高的交互作用,相比与周围的其他点。
大多数的角点附近存在能够被CTCT结合的motif,并且大约有60%~90%的motif表现出趋同的方向。
使用基因组编辑技术翻转motif的方向后,破坏了角点和对应的TAD
短时间的降解CTCF蛋白后也会破坏loop 域

Compartmentalization 是染色质域的形成第二种机制
隔室的定义最开始是在1M的Hi-c中定义的,其中包括超长距离的染色体内的交互与染色体间的交互。这种划分将基因组划分成了两种类型
A compartments包括一些有活性的转录基因、组蛋白标记
B compartments 没有活性的基因和组蛋白标记
在最开始低精度的Hi-c图谱中认为,TADs是嵌套在单个compartments中的,随着精度的提高人们通过对多种有活性和没有活性的compartments进行组合后将染色体分割为6种亚compartments。
TAD + compartment domain: un-nested corner-dot + compartment domain
TAD only: un-nested corner-dot domain only
Compartment domain only: un-nested no-corner-dot compartment domain
Nested subTAD + compartment domain: nested corner-dot compartment domain
Nested subTAD only: nested corner-dot domain only
Compartment domain only: nested no-corner-dot subcompartment domain
三种域之间的比较
通过对 cohesin 进行突变之后:发现compaetments强度增加而TAD与subTAD减少,表明compartments和‘TAD|subTAD’是染色质区域化的两种不同机制
单细胞水平比较TAD和compartment domains
通过显微成像技术,发现单个细胞之间的TAD水平存在差异,也可能是细胞发育状态不一样,TAD水平不同步;但是总体上对boundary的区分还是比较明显的。
在compartments的比较中发现,compartment domains的形成是比较同步的、less random

功能上的差异
最终的问题就回到了结构和功能之间的关系了
Evidence to date indicates that TADs and subTAD loop domains, compared with A/B compartments, exhibit a distinct functional connection to gene regulation.
TAD loop 域影响转录的三种机制
增强子和启动子通过持续性的loop发生直接的交互图a
弱交互,增强子和启动子位于同一个loop但是没有共同定位在边界处图b
错误的靶向了其他基因图c

在一些研究中,对TAD结构域的破坏并没有表现出很多基因表达水平上的改变,可能是由于增强子和基因之间的交互距离本身就不是很远。
总结
了解了Hi-c技术的发展和一些专业术语的
从基因组结构到对应的功能model,loop促进增强子和启动子之间的交互
subTADs与TAD在不同细胞类型,不同发育时期的差异
动物TAD loop的异常往往导致基因表达的异常
TAD loop boundary的差异
recent reports have indicated a role for transposable elements in the formation of cell-type-specific boundaries38,39. Thus, an important area for future inquiry will be to unravel the structural, functional and mechanistic differences among boundaries across length scales
参考
Last updated