经典Hi-C文献
A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping
highlight
这篇文章通过改进了Hi-C的实验后,提高了分辨率,并且得到了以下结果:
将genome中 交互contact domains 的大小定位到185kb左右
根据组蛋白修饰将compartment细分成6个subcompartment
在不同组织中鉴定到了~10000个染色质 loops
loops与基因启动子、增强子有关,并且能够促进基因的表达
在不同细胞和不同物种间,loops有着比较高的保守性
发现大部分loops与compartment的边界重合现象
根据highlight思考
首先相比于之前的Hi-C,suit Hi-C做了哪些改进
Hi-C分析中,怎么计算domain的分辨率
如何划分subcompartment
怎么鉴定loops
Hi-C实验的改进

一代Hi-C技术 dilution Hi-C:
通过将细胞核打碎后,在溶液中进行胶联
使用6碱基的HindIII内切酶
胶联时间比较长
suit Hi-C技术:
在细胞核内进行胶联,减少一定的假阳性
使用4碱基DNA内切酶,酶切效率更高
胶联时间缩短,由7d变为3d
Hi-C中的一些概念和图片

matrix resolution: 在Hi-C热图中人为定义的窗口的大小
map resolution:在基因组中的片段中,80%的位置存在至少1000次交互,这些片段中最小的那个片段长度;一般也就代表了Hi-C实验的最高分辨率了
通过将染色体拆分成固定大小(10K,100K,1M)的连续片段,通过统计两两片段间交互的频率就可以得到一个二维矩阵M~i,j~,i和j当然代表染色体不同的两个位置
interva:是一窜连续的位置,如果两个interva发生了交互,那么在热图中就显示成一个矩形或者正方形
例如图中 第一行是在500KB 的matrix resolution精度下,第二行是在50kb的精度下;其中数字21表示的就是interva的数目
得到contact domain的大小
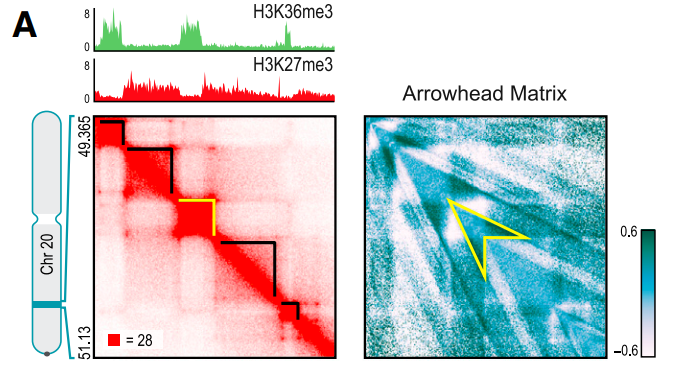
大致就是根据Hi-C构建的交互矩阵结果,通过分析interva,在图中也可以看出热图可以被区分成一个一个小方块。使用Arrowhead algorithm算法,得到这些方块的大小在40Kb~3M之间,中位数大致185kb
根据组蛋白修饰分成6个sub compartment

通过使用随机聚类的算法,将concat Domains分成6个subcompartment,其中A1、A2亚组有更多高表达的基因,活跃型组蛋白修饰H3K36me3, H3K79me2 也很多。
鉴定染色质loops

Such pairs correspond to pixels with higher contact frequency than typical pixels in their neighborhood
通过比较与局部比对的read进行比较,发现显著性的peak,这里的pixels也就对应了一个matrix resolution的大小,与此同时作者开发了一套鉴定染色体loop的流程juicer-tools。
试了一下,感觉软件挺好用。
juicer-tools 教程:https://github.com/BiocottonHub/BioSoftware/wiki/juicer-tools
Loops保守性分析
分析了不同细胞类型和不同物种中保守的loops后,发现在不同细胞中loops大多数是保守的,在老鼠和人中大约有50%的loops是保守的
loops与启动子、增强子
通过对基因启动子进行分类,分层在loop区域和不在loop区域。发现启动子在loop区域的表达水平比没有loop的高出6倍之多
同时分析了人类和老鼠中的一个基因SELL,在人类中SELL存在loop(左图,蓝圈);而老鼠中不存在loop;人类中SELL是表达的,而老鼠中不表达。
loops可能在3维空间上使得启动子和增强子靠近促进基因表达

参考
A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping
Last updated