AS统计
统计PB支持的AS
提取那些有PacBio支持的AS事件,并且去除了Scaffold,提取剪切事件所发生的转录本;
并且将那些剪切位置相差几个bp的AS合并了
### 对于IR事件发生在有重叠的坐标区域时,把对应的区域进行合并
python arrangeAS.py -AS ~/work/Alternative/result/Gr_result/CO41_42_result/11_AS/end_splice.txt -r ~/work/Alternative/data/Gr_genome/Graimondii_221_v2.1.gene.gtf -p ~/work/Alternative/result/Gr_result/CO41_42_result/07_annotation/merge.gtf -o D5_AS.txt统计AS的种类和数目
事件数目
基因组
IR
ES
AltA
AltD
AltP
Other
A2
21575
2352
5847
4472
4394
4269
D5
18345
2229
5350
3845
4185
6302
TM1
32838
3723
9749
8252
7358
7915
At
16123
1850
4878
4011
3621
3882
Dt
16715
1873
4871
4241
3737
4033
基因数目
基因组
IR
ES
AltA
AltD
AltP
Other
A2
9138
1499
3147
2614
2200
1881
D5
7463
1443
2900
2295
2214
2285
TM1
13573
2399
5414
4740
3780
3308
At
6656
1210
2709
2345
1872
1672
Dt
6917
1189
2705
2395
1908
1636
IR和其他AS的关系
出现IR的地方同时也似乎容易出现AltA、AltD等情况,分析先有IR还是先有AltA、AltD;
获取剪切事件所在的基因组坐标而不是内含子坐标
提取既有IR又有AltA、AltD的基因出来;分析AltA与IR之间是否有着某种联系
在存同时存在IR与AltA、AltD事件时;AltA、AltD与IR之间间隔小于2bp碱基
将近有27%左右的AltA、AltD与IR的事件的坐标是相差一个碱基的差异
基因组
AltA
AltD
TM1
1015/3675
909/3385
A2
493/2032
538/1876
D5
527/1840
508/1638
鉴定保守的AS
保守的IR
基因组
存在IR基因
保守基因
保守事件
基因组1
基因组2
A2 At
3570
2268
3827
9735
9580
D5 Dt
3295
1901
2956
9142
8828
A2 D5
3609
2033
3188
9652
9708
At Dt
2911
1734
2737
7944
7871
保守的ES
基因组
存在IR基因
保守基因
保守事件
基因组1
基因组2
A2 At
299
189
200
576
543
D5 Dt
285
163
186
569
573
A2 D5
262
128
138
492
481
At Dt
250
141
151
474
478
保守的AltA
基因组
存在IR基因
保守基因
保守事件
基因组1
基因组2
A2 At
891
462
523
2030
1960
D5 Dt
835
413
473
1919
1791
A2 D5
765
349
389
1704
1746
At Dt
820
395
448
1792
1836
保守的AltD
基因组
存在IR基因
保守基因
保守事件
基因组1
基因组2
A2 At
659
348
385
1380
1358
D5 Dt
640
312
353
1344
1432
A2 D5
544
224
247
1055
1020
At Dt
662
344
396
1414
1456
uniq IR在另外一个基因组的坐标
这个uniq的IR在另外一个基因存在三种情况:
在另外一个基因组是intron,持续被剪切的状态
在另外一个基因组变成exon,已经进化成exon了
提取基因的固定长度的k-mer序列,使用bwa进行比对,获得uniq IR在另外一个基因组上的坐标;考虑到发生uniq IR的内含子在两个基因组中的长度可能会不一样,同时结合两端FEST序列进行一次锚定
使用k-mer方法进行实验
首先提取与uniq IR一样长的k-mer片段,实验wu-Blast进行比对,筛选必读长度和IR一样的,并且得分最高的
先进行的是k-mer方法,之后又使用两端FEST方法;看两种方法的交集怎么样
由于FEST取的是左右300bp,因此假阳性情况会比较多,尽量与k-mer的数据为准
将保守的与uniq的合并,统计IR在两个基因组的情况
统计这个区域是外显子、或者内含子
把所有的区域计算一下read的覆盖情况
将这段区域,与PacBio注释文件、参考基因组注释文件进行比较;判断它是否一直是以外显子的形式、或者一直以内含子的形式进行转录;还有一种复杂的情况是Ohter类
提取参考基因组和PacBio每个转录本的注释信息;将这段区域与转录本的注释信息进行比较;如果与内含子或者exon的交集,达到这个片段的90%以上,就认为这个区域是被注释为内含子或者外显子;
如果在一个转录本中被注释为exon,另外一些被注释成intron;则认为发生了IR事件。最后统计在一个基因中所有转录本的情况;如果都满足注释为内含子或者都瞒住注释为外显子,就认为该区域为组成型内含子或组成型外显子
外显子化
内含子化
Other
r1 p1对应2号文件的第一列
统计比例
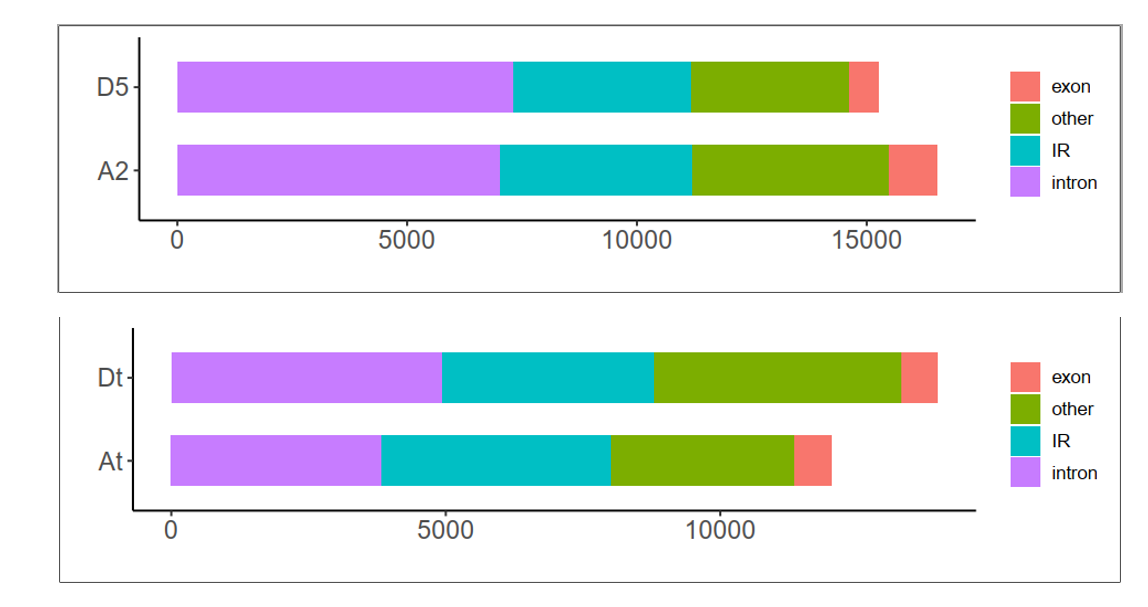
画一个堆积的条形图:
A2 vs At
IR保守率:
基因组
外显子化
内含子化
other
保守
A2发生IR
1091
7650
3602
4221
At发生IR
686
4170
2958
4221
D5 vs Dt
基因组
外显子化
内含子化
other
保守
D5发生IR
774
7342
3278
3875
Dt发生IR
808
4912
4366
3875
A2 vs D5
基因组
外显子化
内含子化
other
保守
A2发生IR
805
6187
4002
3766
D5 发生IR
480
5181
2725
3766
At vs Dt
基因组
外显子化
内含子化
other
保守
At发生IR
730
5486
2616
3234
Dt发生IR
777
5503
2765
3234
,大部分变成了intron,少部分变成exon;四倍体中的IR大部分是由二倍体中的intron转变而来的。
并且在A2中有 1049/16545 的IR发生了外显子化; D5中774/15269 的IR发生外显子化。
计算类型PIR值的变化
外显子化的IR
变成Intron的IR
保持IR不变的IR
可以看出,发生外显子化的IR,它的PIR值很高;而发生intron化的内含子PIR值很低;
由于不同基因组在比较的时候,总read数目存在一个差异,统一将TM1再除以1.35倍
PIR值:比对到intron的read/(intron+两端read)
外显子化的IR、与内含子化的IR在PIR值上的差异;多倍化后外显子化的IR,PIR值显著性的增加
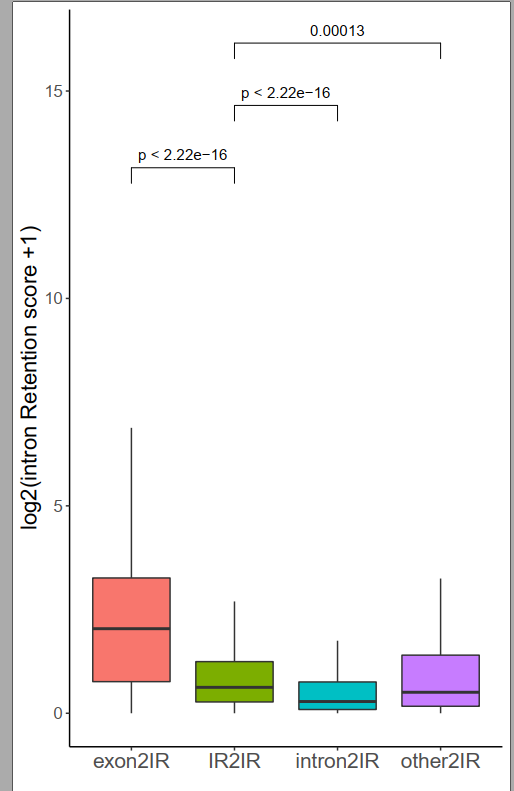
比较PIR值的差异:
由exon 变成IR状态,PIR值仍旧保持一个较高的值,说明这个位置不容易被剪切掉
原先是exon,变成IR;仍旧是发生IR占据主导地位;当然也有剪切类型占据主导地位的
原先是intron,变成IR
原先是IR,变成IR
原先是other,变成IR
IR变成intron的过程,PIR的值不减少,反而有所增加;可能是有些IR没有被鉴定到;或者存在AltA、AltD的情况导致的由于多倍化物种在比对的时候,有问题因此在对A2和At间进行比较的时候,误差感觉比较大
在同一个基因组内进行比较
比如在A2中都是IR,为啥有的多倍化之后变成了外显子、内含子、IR、Other
在At中都是IR,不同来源的IR,在剪切效率上同样不同
Intron Ration score: 比对到内含子上的read数/内含子长度;再把read总数给标准化
通过对A2中的IR分析发现,大部分的IR在多倍化之后变成了内含子;根据分类发现外显子化的内含子有着最高的IR score值
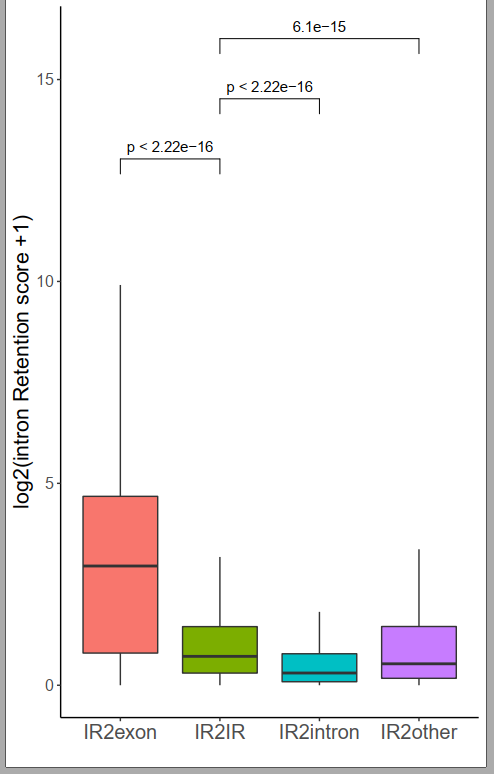
通过对At中的IR分析发现,大部分的IR在二倍体中同样是IR;同样有些在二倍体中是exon;在四倍体中变成IR;并且这些内含子化的exon在mRNA中仍旧有较高比例的保留
看一下外显子化的IR有多少交集,并且进行GO富集分析
这种外显子化、内含子化,可能起着调控基因表达的作用
A2多倍化为At时,有一个IR发生了外显子化;而D5与Dt始终是外显子;表达量增加
A2多倍化At时,有一个外显子发生了内含子化;而D5与Dt始终是外显子;表达量减少
A2多倍化过程中外显子化的IR:801个,其中有389个IR在D5中是外显子
D5多倍化过程中外显子化的IR: 389个,其中有173个IR在A2中是外显子
二倍体祖先基因组,IR外显子化
四倍体IR由外显子转化而来的IR
At基因组中由exon转变为IR:552个,其中有233个在Dt中是exon的状态
Dt基因组中由exon转变为IR:492个,其中有308个在At中是exon的状态
GO富集分析
At中的GO
A基因组外显子化的基因、从外显子转变成IR的基因;因为即使变成IR之后PIR值也还是很高的
Dt中的GO
亚基因组特异性的AS
At与Dt进行比较的时候,例如At中存在IR、Dt中不存在IR;并且At中那个IR的地方有很多read覆盖,而Dt中几乎没有read覆盖
提取At中发生IR的IRscore ,Dt中没有发生IR的IR score;存在显著性差异
At中发生IR、Dt中没有发生IR;并且发生IR的IR score值大于0.1,没有发生IR的值小于0.1
排除那些由于PacBio没检测到的IR
A2与D5进行比较
Last updated