01三代测序Iso-seq
PacBio三代全长转录组测序
分析流程
https://github.com/PacificBiosciences/IsoSeq/blob/master/README_v3.1.md

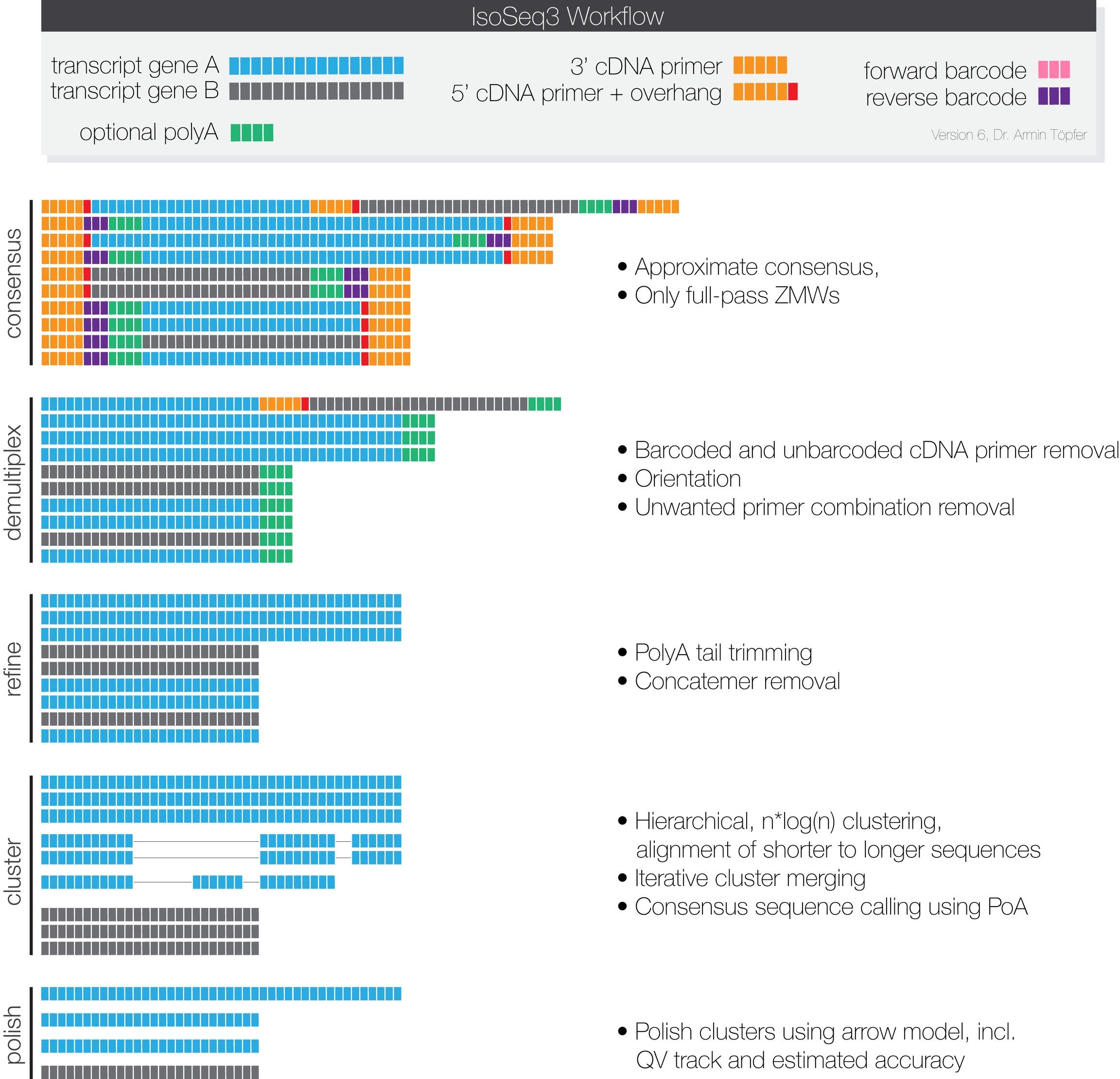
这张图片形象的展示了每一步测序数据的变化
1.安装SMART软件
从网站https://www.pacb.com/support/software-downloads/下载SMART-Link软件
--rootdir 指定安装路径
--smrttools-only 只安装命令行工具,反正服务器里你图形界面也看不到
版本升级,下载升级版本的安装文件,然后只需要在安装的命令上面加个参数就行
之后将安装的命令添加到环境变量中
2.使用CCS对原始数据进行过滤
noPolish 不会对数据进一步的过滤
-minPasses 最低的通过值
minLength 获取的draft consensus最短长度,用于下一步的分析
maxLength 最长的长度
minSnr 移除包含delete的SNP
-minPredictedAccuracy 最小精度0.8
--logFile 记录日志文件
-reportFile 报告处理的文件
maxDropFraction Maximum fraction of subreads dropped by polishing
3.对转录本进行无参考基因组的归类
引物文件是固定的
3.1 去除引物
lima output.bam primers.fasta demux.ccs.bam --isoseq --no-pbi -j 线程数 --min-length 300
3.2 去除full length 的噪音remove polyA tails
3.3 聚类
` isoseq3 cluster movie.flnc.bam unpolished.bam ·
:warning:如果想要比较同源基因之间的差异的话,这一步可以不做
3.4打磨 polich
isoseq3 polish -j 20 unpolished.bam input_subreads.bam polished.bam
4.比对到参考基因组
4.1 软件安装
4.2 建立参考基因组
4.3 将全长转录本比对到参考基因组
-f 输出文件为sam格式
-t 指定线程数目
-n 设置比对的类型,为0可以鉴定嵌合基因
4.4 根据SNP数据区分多倍体reads
对于mapping状态可分为以下几类:
M:alignment match (can be a sequence match or mismatch)
表示read可mapping到第三列的序列上,则read的碱基序列与第三列的序列碱基相同,表示正常的mapping结果,M表示完全匹配,但是无论reads与序列的正确匹配或是错误匹配该位置都显示为M
I:insertion to the reference
表示read的碱基序列相对于第三列的RNAME序列,有碱基的插入
D:deletion from the reference
表示read的碱基序列相对于第三列的RNAME序列,有碱基的删除
N:skipped region from the reference
表示可变剪接位置
P:padding (silent deletion from padded reference)
S:soft clipping (clipped sequences present in SEQ)
H:hard clipping (clipped sequences NOT present in SEQ)
clipped均表示一条read的序列被分开,之所以被分开,是因为read的一部分序列能匹配到第三列的RNAME序列上,而被分开的那部分不能匹配到RNAME序列上。
"="表示正确匹配到序列上
"X"表示错误匹配到序列上
5.TaMa将很相似的转录本合并去冗余
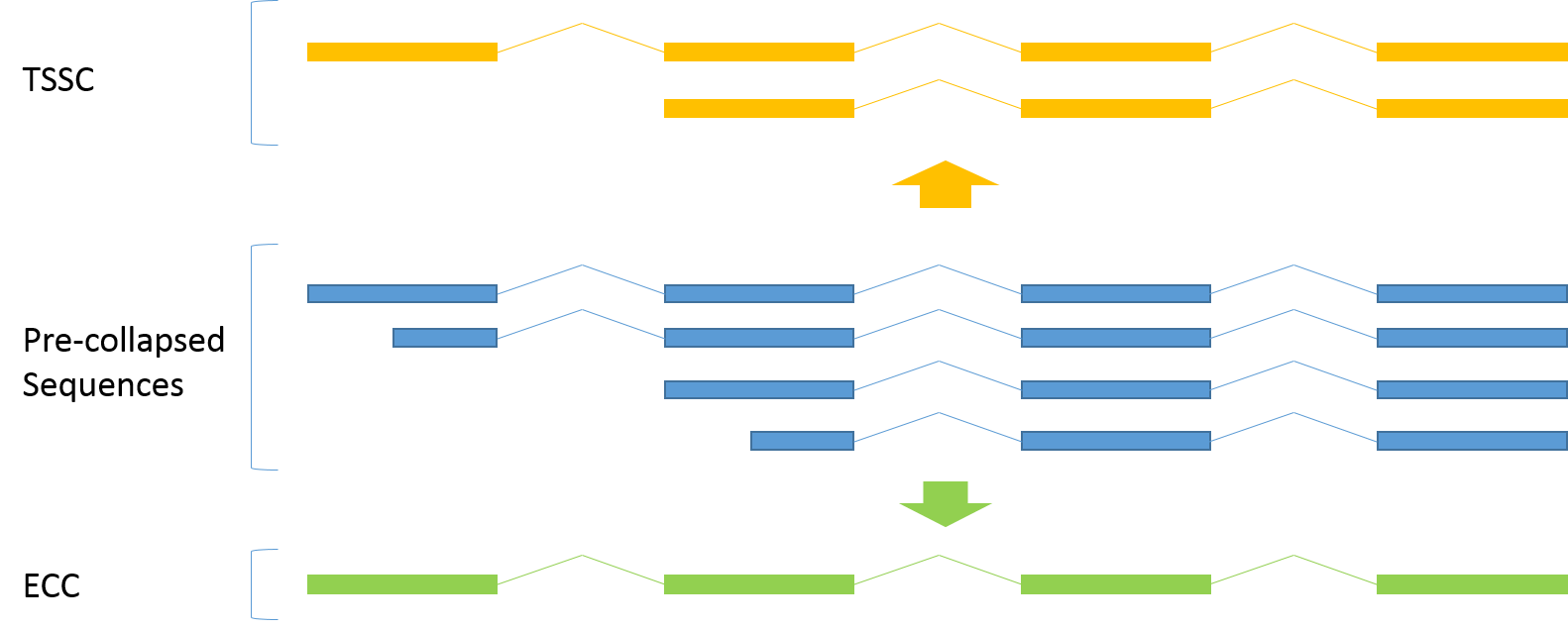
这个过程很复杂,图中就有两种可能的合并方式:
Transcription Start Site Collapse
Exon Cascade Collapse
具体可以查看这篇文献 https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-017-3691-9
5.1 下载和安装
运行tama_collapse.py 脚本
每个参数的详细说明 https://github.com/GenomeRIK/tama/wiki/Tama-Collapse
6.Cupcake去除冗余,这个步骤和5是一样的 推荐这个流程
6.1 安装Cupcake软件,这个流程适合依赖于python2的cupcake,应该克隆对应的Py2_v8.7.x. 分支
首先得安装cogent环境 https://github.com/Magdoll/Cogent/wiki/Installing-Cogent#conda
6.2 安装依赖于python3.7环境的cupcake
参考 https://github.com/Magdoll/cDNA_Cupcake/tree/master
6.3 具体的使用方法
--input 输入文件
--fq 指定输入文件为fastq
-s Gmap输出后的sam文件经过sorted
-o 输出文件前缀,当然也可以加目录,直接输出到对应目录下
-c 最小覆盖度
-i 相似度
--dun-merge-5-shorter 5‘端的read由于测序的原因可能是真是存在差别,也可能是冗余;跟建库方式有关;因为设计引物数利用ployA的,所以5’端的序列可能没有完全扩到
过滤因为5’端测序的误差,导致冗余没有完全去除
7.与已有的注释信息进行比较
https://github.com/TomSkelly/MatchAnnot
报错
8.Alternative splice.py脚本进行分类
:warning:alternative_splice.py脚本中使用的参考基因组的gtf文件还需要使用awk,进行转化
输出文件
暂时就更新到这里了~~~
鉴定可变剪切spladder
参考文档 https://spladder.readthedocs.io/en/latest/installation.html
在Ancona中安装这个软件
进行可变剪切的鉴定
参考
完整的分析 流程 https://github.com/GenomeRIK/tama/wiki
PacBio官方SMART软件使用说明V8版本的 single molecular real-time
https://www.pacb.com/wp-content/uploads/SMRT-Tools-Reference-Guide-v8.0.pdf
https://www.cnblogs.com/RyannBio/p/9598340.html
GMap软件 http://research-pub.gene.com/gmap/
samtools输出文件格式 https://blog.csdn.net/genome_denovo/article/details/78712972
全长转录本分类https://github.com/GenomeRIK/tama/wiki/Tama-Collapse
去除冗余之后的分析流程 https://github.com/PacificBiosciences/IsoSeq_SA3nUP/wiki/What-to-do-after-Iso-Seq-Cluster%3F
Last updated