第7个结果
各个剪切事件并不是显著性的差异
ES数据核实一下
同源基因之间的poly A的差异
组成型intron与剪切intron之间长度差异
同源基因在各个棉种的比较中AS的差异
同源基因间表达的差异与保守度
各个基因组中TE的插入
处理A2的原始数据
参考
AS数据的处理,由于D5基因组相比A2基因组,注释的转录本更多,这里看一下只考虑PacBio测序得到的与参考基因组进行比较的结果
for i in A2 D5 TM-1; do for j in IntronR AltA AltD AltP ExonS Other; do printf ${j}"\t"; grep ${j} ../${i}/end_third|grep PB|wc -l; done; done
和对应的基因数目
for i in A2 D5 TM-1; do for j in IntronR AltA AltD AltP ExonS Other; do printf ${j}"\t"; grep ${j} ../${i}/end_third|grep PB|cut -f2|sort|uniq|wc -l; done; done
基因组
IR
AltA
AltD
AltP
ES
Other
A2
28078
5199
4135
1823
2276
1435
D5
22226
3257
2434
1131
1719
1039
TM-1
40354
7823
6854
4665
3071
2539
At
19869
3868
3341
2301
1531
1197
Dt
20436
3946
3505
2363
1540
1342
看看公司跑的数据,最后就使用这个数据了
鉴定可变剪切的原理:
将PacBio测得的isoform比对到参考基因组,获得isform 注释信息
将PacBio isoform信息与参考基因组组中原有的注释信息进行合并
使用脚本,根据合并后的注释信息中的基因结构信息,提取对应剪切事件和发生的位置
由于A2基因组中原有的注释信息里没有不同转录本的注释信息,并且PacBio测序只测了leaf这一个组织,所以在与A2中原有的注释信息合并后,也不能完全的代表所有的转录本的注释信息
而D5和TM-1基因组中有不同转录本的注释信息,因此在根据基因结构去鉴定AS的时候,有有一些差异。
为了减少这个带来的差异,我在各个棉种中选择那种只被PacBio isoform支持的AS
没有统计scaffold上的AS事件
AS事件数目
基因组
IR
AltA
AltD
AltP
ES
Other
A2
36574
5847
4472
4394
2352
4269
D5
30379
5350
3845
4185
2229
6302
TM-1
51453
9749
8252
7358
3723
7915
At
25386
4878
4011
3621
1850
3882
Dt
26067
4871
4241
3737
1873
4033
gene 数目
基因组
IR
AltA
AltD
AltP
ES
Other
A2
9139
3147
2614
2200
1499
1881
D5
7466
2900
2295
2214
1443
2285
TM1
13574
5414
4740
3780
2399
3308
At
6657
2709
2345
1872
1210
1672
Dt
6917
2705
2395
1908
1189
1636
提取FESTs序列
blast分析保守性
相似度80% ,覆盖度80%以上 e-value 1e-5
统计每种AS对应的比例
保守IR事件
存在保守事件基因的比例:
再计算每个基因,AS事件的保守程度:
保守基因的比例:
2425/7142 33.95%
2274/6594 34.48%
31.91%
62.59%
保守事件的比例:
4789/36144 *2 26.50%
26.95%
24.14%
50.14%
比较
基因对数
事件数
保守基因对数
保守事件数没乘2
A2 vs At
7142
36144
2425
4789
D5 vs Dt
6594
32642
2274
4398
A2 vs D5
7218
35364
2303
4269
At vs Dt
2911
13551
1822
3397
保守ES事件
保守基因的比例:
31.10%
32.11%
23.28%
56.40%
保守事件的比例:
33.85%
36.47%
26.42%
65.40%
比较
基因对数
事件数
保守基因对数
保守事件数没乘2
A2 vs At
598
1152
186
195
D5 vs Dt
570
1146
183
209
A2 vs D5
524
984
122
130
At vs Dt
250
474
141
155
保守AltA事件
保守基因的比例:
55.22%
48.02%
41.05%
45.61%
保守事件的比例:
48.47%
48.25%
41.08%
45.98%
比较
基因对数
事件数
保守基因对数
保守事件数没乘2
A2 vs At
891
2030
433
492
D5 vs Dt
835
1919
401
463
A2 vs D5
765
1704
314
350
At vs Dt
820
1792
374
412
保守AltD事件
保守基因的比例:
49.92%
46.88%
40.26%
46.22%
保守事件的比例:
52.32%
51.19%
44.55%
49.36%
比较
基因对数
事件数
保守基因对数
保守事件数没乘2
A2 vs At
659
1380
329
361
D5 vs Dt
640
1344
300
344
A2 vs D5
544
1055
219
235
At vs Dt
662
1414
306
349
比较不同的AS类型,保守程度是否存在差异
计算保守基因对,的AS事件保守程度
不同剪切事件在多倍化的过程中,保守的程度不一样,其中ES最保守,而IR保守性最低
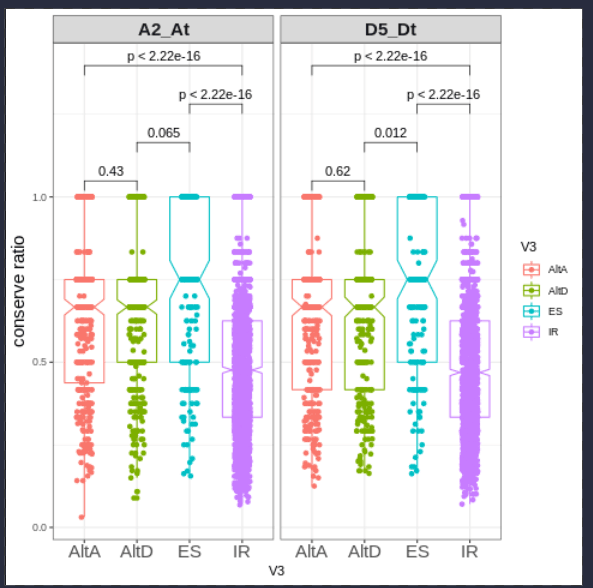
同一类剪切事件,在不同基因组的比较中的保守程度差异
A2 vs At 叫保守,D5 vs Dt叫保守;
A2 vs D5叫并行分化,At vs Dt叫趋同进化
不同剪切事件的长度差异
组成型的内含子
保守的剪切事件对应的intronR
不保守的剪切事件对应的intronR
组成型的外显子
发生ES的外显子
保守的ES
对多倍化过程中的剪切事件进行分类
parallel conserve
A2与D5中保守,在At与Dt中同样保守
A2与D5中不保守,但是在At与Dt中保守;A2 At或者D5 Dt中存在保守
bias conserve
A2与D5中保守,但是在At与Dt中不保守;A2 At或者D5 Dt中存在保守
A2与D5中不保守,在At与Dt中同样不保守;但是在A2 At或者D5 Dt中是保守的
四倍体内同源基因更多的发生分化
分析A2与D5中保守的剪切事件,在At、Dt中的保守情况 1. A2 D5中保守,在At与Dt中同样保守 2. A2 D5中保守,在At与Dt中不保守
A2和D5基因组分别是一个独立的个体,存在一些保守的AS,维持正常的生命活动;
剪切机制的存在往往是为了让植物能够快速响应外界环境的变化,在正常的环境中这种机制的存在往往是能量的浪费,因此在四倍体中,发现大多数原来在二倍体内保守的剪切事件,在四倍体内发生了分化,只在At中存在,或者只在Dt中存在,这两种比例各占一半
A2和D5作为一个独立的个体,因此具有一些保守的剪切事件;而剪切事件往往是一种能量的浪费;在四倍体内只需要某一个基因组存在对应的AS即可
类型
二倍体内保守
四倍体内保守
四倍体内不保守
IR基因
2303
536
1154
IR事件数
4269
838
1697
ES基因
122
21
46
ES事件
130
22
47
AltA基因
314
48
121
AltA事件
350
51
127
AltD基因
219
37
77
AltD事件
235
40
80
绘制图形
在画一个维恩图,A中保守的,D中保守的,At、Dt中同样保守的比例
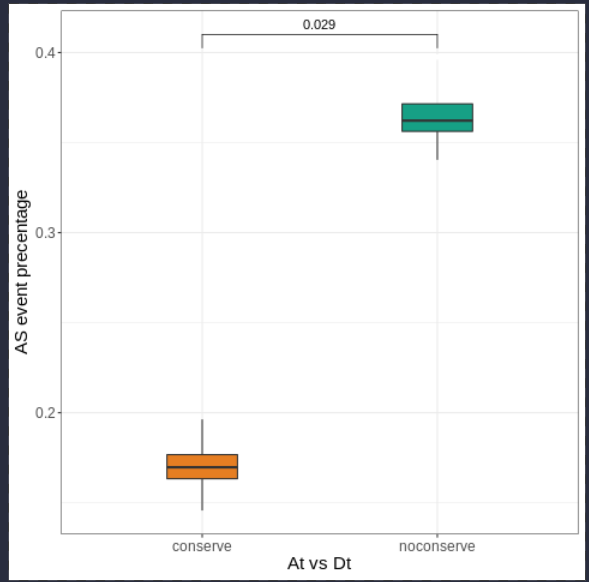

看剪切事件在两个亚基因组间是否存在偏向性
只在A基因组中保守的剪切事件,占A2和At中保守事件的比例
只在D基因组中保守的剪切事件
事件类型
只在A保守的占A中所有保守比例
只在Dt中保守的占D中所有保守的比例
IR
3623/4461
3189/4027
ES
164/176
154/186
AltA
394/445
358/409
AltD
287/327
270/310
分析每个基因组中特异剪切事件对应的GO
在A基因组中特异的AS对应的基因富集的GO
在D基因组中特异的AS对应的基因富集的GO
A、D中保守的AS对应的基因富集的GO
分析可变剪切对基因表达式水平的影响
At与Dt中存在保守事件的基因
At与Dt中仅仅只有一个亚基因组中存在保守事件的基因
存在保守事件的同源基因对间的差异表达情况
不存在保守事件的同源基因对的差异表达情况
Divergence of duplicated genes by repeated partitioning of splice forms and subcellular localization
剪切事件在多倍化过程中的变化
在二倍体中存在的事件,并且在四倍体中At、Dt保守;多倍化之前就存在的事件
在二倍体中不存在的事件,但是在四倍体中保守;多倍化后新产生的事件
参考
Last updated