手把手教你搭建JBrowse-初始化应用
JBrowse是GMOD开源项目中的一个基因组浏览器,所谓的基因组浏览器,字面意思就可以理解成一个网页应用。可能每个生物信息分析的同学电脑内都会安装一个IGV,用于对高通量测序数据的可视化。其实JBrowse也就是相当于把IGV,搬到了服务器内,把庞大的测序数据存储在服务器内,借助于快速发展的web技术;使得我们能够通过仅仅发送一个网页链接就能够与他人共享或者讨论课题。
本文搭建JBrowse应用访问链接: http://cotton.hzau.edu.cn/tools/jbrowse/?data=data/Ghirsutum_genome_HAU_v1.1

配置文件
JBrowse支持两种配置文件:
JSON格式
textual格式
其中JBrowse的全局配置文件jbrowse.conf采用textual格式进行配置,具体到特定的数据例如 基因组序列,BAM文件的配置信息时采用textual格式;具体到某种数据的显示时采用JSON进行配置
入门文章也是使用简化的配置:https://jbrowse.org/docs/minimal.html
主要讲一下tracks.conf这个textual类型的配置文件
[tracks.refseq]后面的refseq表示轨迹的名称,之后再WEB上的选项上会出现urlTemplate表示轨迹文件在的路径,相对于当前配置文件的路径storeClass轨迹的类型包括fasta、vcf、BAMtype字段也差不多
文件建立索引
建立索引的目的是让JBrowse能够快速的找出目标位置的序列信息
fasta文件建立索引
使用samtools中的子程序faidx建立基因组索引,这样在JBrowse中就可以快速的导出指定片段的fasta序列信息
配置文件tracks.conf,告诉JBrowse;基因组文件在哪里
gff文件索引
使用GFF3文件构建索引,在建立索引之前需要对GFF3文件进行排序,安装染色体和位置进行排序
在排序完成后进行压缩和建立索引;使用tabix软件进行;
tabix安装参考 https://www.jianshu.com/p/b6f885fc8b5f
在tracks.conf文件中添加配置信息
BAM文件索引
BAM文件同样需要使用samtools按照染色体位置排好序,再建立索引;再JBrowse更新到v1.15.0版本后支持了CRAM格式文化,这种格式相比于BAM格式更加节省存储空间
JBrowse可以对BAM文件进行两种展示:
参考 BAM两种展示形式 https://jbrowse.org/docs/tutorial_classic.html#next-gen-read-track-types
set type = Alignments2 ;显示单个read的比对情况
type = SNPCoverage ;显示某个区域read的覆盖情况
在tracks.conf文件中添加配置信息
配置完成后的目录结构
定制轨迹
修改参考序列轨迹
参考修改 序列轨迹 https://jbrowse.org/docs/reference_sequence.html#reference-sequence-display-order
可以通过修改CSS来改变5种碱基 A T C G N显示的颜色
修改 CanvasFeatures
使用flatfile-to-json.pl脚本进行转换
信号转发
现在访问http://cotton.hzau.edu.cn/tools/jbrowse/?data=data/Ghirsutum_genome_HAU_v1.1 就可以显示出内网中的JBrowse
由于我把JBrowse放在了内网机器上,于是需要使用外网机器的Appache将内网信号转发一下;在转发后客户端找不到对应的CSS和JS文件;于是使用webpack重新打包了一下源代码;查看了一下webpack的配置文件,发现可以通过配置环境变量JBROWSE_PUBLIC_PATH的值来控制打包后的资源检索URL
修改打包后的公共资源请求路径,打包后的文件会在jbrowse/dist目录下
如果没有yarn,使用npm进行全局安装 npm i yarn -g
修改@/jbrowse/index.html中资源路径;其实就加个转发路径就行
sed -i 's/href=\"/href=\"\/tools\/jbrowse\//g' index.html
sed -i 's/src=\"/src=\"\/tools\/jbrowse\//g' index.html
效果展示
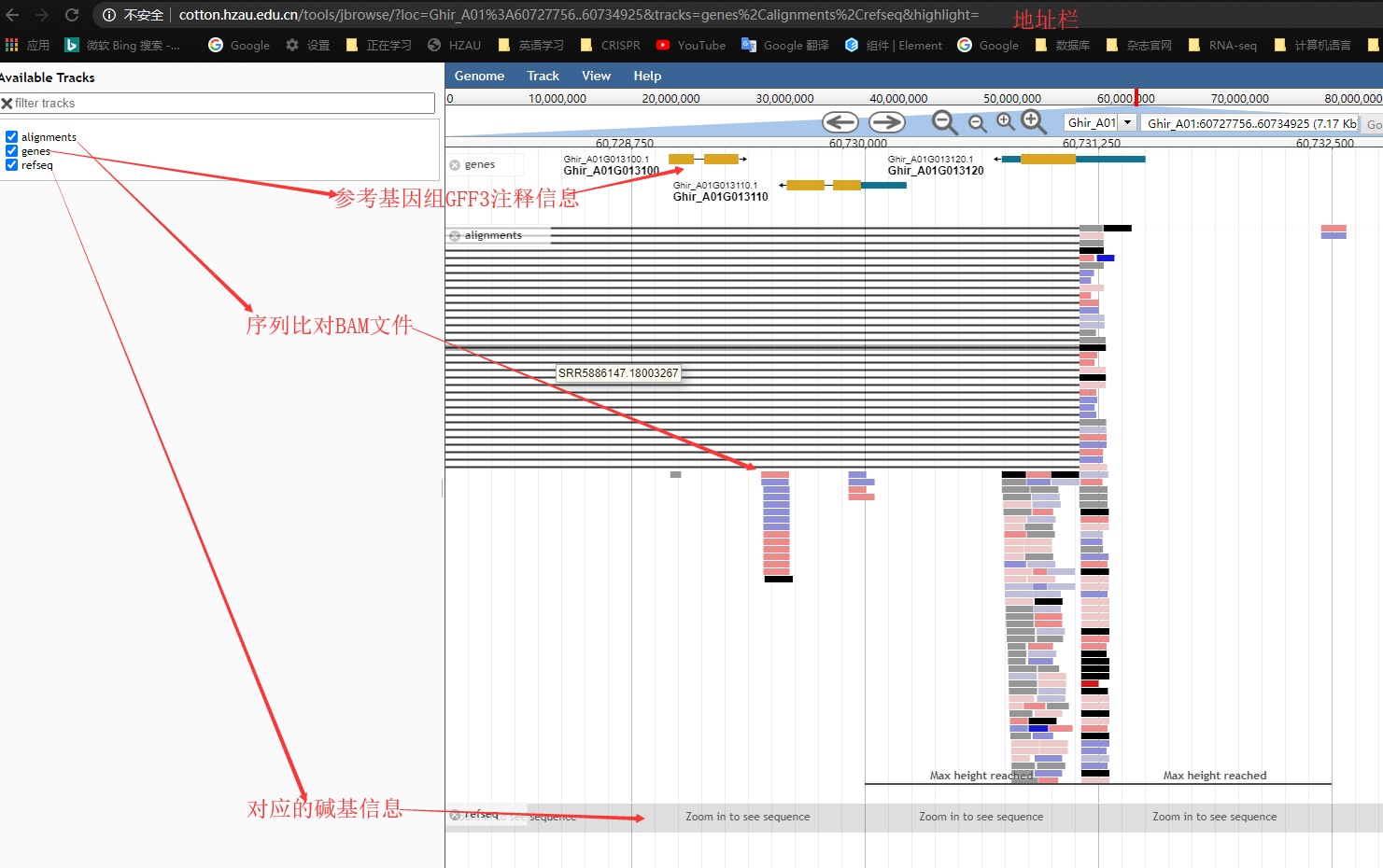
参考
Last updated